El Capítulo 13, “Análisis de Varianza Combinado”, en Experimentación en Agricultura, junta varios análisis descritos en secciones previas.
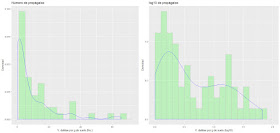 |
A la
izquierda, número de propágalos cuantificados durante muestreo (no.); a la derecha,
números del muestreo transformados con log10(no. + 1).
|
También, da una excelente exposición sobre análisis e interpretación de datos recopilados periódicamente, ej. Respuesta, en altura de planta a aplicaciones de fertilizantes; ganancia de peso en cerdos alimentados con diferentes dietas. Datos que cómodamente
son recolectados diaria-, semanal-, o mensualmente, con intervalos sucesivos, desde
el inicio hasta el final del experimento.
Para aliviar sobrecarga de códigos, rebanamos el Capítulo en 5 entregas: ANOVA combinado (1) — un muestreo; ANOVA combinado (2) — mediciones temporales repetidas; ANOVA combinado (3) — diferentes épocas-años; ANOVA combinado (4) — diferentes localidades; ANOVA combinado (5) — experimento de larga duración.
 |
|
En los siguientes
códigos, ofrecemos opciones para representar y calcular el mismo gráfico o
resultado. Lo hacemos para archivar la plétora de alternativas existentes.
Códigos en R
#***********************************************************
# EXPERIMENTACIÓN EN AGRICULTURA
# Fernández Escobar, R.; Trapero, A.; Domínguez, J. 2010
# CAPÍTULO 13: ANÁLISIS DE LA VARIANZA COMBINADO
# (páginas 167 - 172)
#************************************************************
# Importar datos
datos <- read.csv("C:/Users/Administrator/Desktop/tabla 13.1.csv", header=TRUE)
attach(datos); datos#### Crear y etiquetar variables en datos "Propágalos"
# tratamientos etiquetados
TRAT <- factor(TRAT, levels = c(0,1,2), labels = c("Sin solarizar", "Solarizado 1", "Solarizado 2"))
REP <- factor(REP)
MUES <- factor(MUES)
datosID <- data.frame(TRAT, REP, MUES, PROP); datosID# transformación a log base 10
LOPROP <- log10(PROP+1)
datosT <- data.frame(datosID, LOPROP); datosT## TRAT REP MUES PROP LOPROP
## 1 Sin solarizar 1 1 18.4 1.28780173
## 2 Sin solarizar 1 2 19.8 1.31806333
## 3 Sin solarizar 1 3 23.6 1.39093511
## 4 Sin solarizar 1 4 60.2 1.78675142
## 5 Sin solarizar 1 5 34.5 1.55022835
## 6 Sin solarizar 1 6 15.7 1.22271647
## 7 Sin solarizar 2 1 28.3 1.46686762
## 8 Sin solarizar 2 2 65.4 1.82216808
## 9 Sin solarizar 2 3 23.1 1.38201704
## 10 Sin solarizar 2 4 34.5 1.55022835
## 11 Sin solarizar 2 5 9.6 1.02530587
## 12 Sin solarizar 2 6 16.4 1.24054925
## 13 Sin solarizar 3 1 13.4 1.15836249
## 14 Sin solarizar 3 2 12.0 1.11394335
## 15 Sin solarizar 3 3 7.1 0.90848502
## 16 Sin solarizar 3 4 15.0 1.20411998
## 17 Sin solarizar 3 5 9.6 1.02530587
## 18 Sin solarizar 3 6 45.3 1.66558099
## 19 Sin solarizar 4 1 16.5 1.24303805
## 20 Sin solarizar 4 2 8.1 0.95904139
## 21 Sin solarizar 4 3 12.7 1.13672057
## 22 Sin solarizar 4 4 15.8 1.22530928
## 23 Sin solarizar 4 5 6.6 0.88081359
## 24 Sin solarizar 4 6 33.2 1.53402611
## 25 Solarizado 1 1 1 0.6 0.20411998
## 26 Solarizado 1 1 2 1.5 0.39794001
## 27 Solarizado 1 1 3 1.1 0.32221929
## 28 Solarizado 1 1 4 6.2 0.85733250
## 29 Solarizado 1 1 5 0.5 0.17609126
## 30 Solarizado 1 1 6 0.2 0.07918125
## 31 Solarizado 1 2 1 0.7 0.23044892
## 32 Solarizado 1 2 2 2.7 0.56820172
## 33 Solarizado 1 2 3 1.2 0.34242268
## 34 Solarizado 1 2 4 4.4 0.73239376
## 35 Solarizado 1 2 5 7.3 0.91907809
## 36 Solarizado 1 2 6 0.5 0.17609126
## 37 Solarizado 1 3 1 7.0 0.90308999
## 38 Solarizado 1 3 2 3.6 0.66275783
## 39 Solarizado 1 3 3 0.8 0.25527251
## 40 Solarizado 1 3 4 0.5 0.17609126
## 41 Solarizado 1 3 5 1.2 0.34242268
## 42 Solarizado 1 3 6 0.9 0.27875360
## 43 Solarizado 1 4 1 2.3 0.51851394
## 44 Solarizado 1 4 2 0.8 0.25527251
## 45 Solarizado 1 4 3 3.7 0.67209786
## 46 Solarizado 1 4 4 1.9 0.46239800
## 47 Solarizado 1 4 5 6.5 0.87506126
## 48 Solarizado 1 4 6 1.5 0.39794001
## 49 Solarizado 2 1 1 2.8 0.57978360
## 50 Solarizado 2 1 2 1.2 0.34242268
## 51 Solarizado 2 1 3 0.6 0.20411998
## 52 Solarizado 2 1 4 0.9 0.27875360
## 53 Solarizado 2 1 5 0.0 0.00000000
## 54 Solarizado 2 1 6 0.3 0.11394335
## 55 Solarizado 2 2 1 0.0 0.00000000
## 56 Solarizado 2 2 2 0.2 0.07918125
## 57 Solarizado 2 2 3 0.4 0.14612804
## 58 Solarizado 2 2 4 2.6 0.55630250
## 59 Solarizado 2 2 5 0.3 0.11394335
## 60 Solarizado 2 2 6 0.5 0.17609126
## 61 Solarizado 2 3 1 1.0 0.30103000
## 62 Solarizado 2 3 2 2.7 0.56820172
## 63 Solarizado 2 3 3 1.8 0.44715803
## 64 Solarizado 2 3 4 0.5 0.17609126
## 65 Solarizado 2 3 5 0.0 0.00000000
## 66 Solarizado 2 3 6 0.8 0.25527251
## 67 Solarizado 2 4 1 0.5 0.17609126
## 68 Solarizado 2 4 2 1.6 0.41497335
## 69 Solarizado 2 4 3 0.4 0.14612804
## 70 Solarizado 2 4 4 0.2 0.07918125
## 71 Solarizado 2 4 5 0.2 0.07918125
## 72 Solarizado 2 4 6 0.0 0.00000000attach(datosT)#### Estadística Descriptiva y Gráficos
source("C:/Users/Administrator/Desktop/summarySE.r") # Función summarySE
# sin transformar
summarySE(datosT, measurevar = "PROP", groupvars = c("TRAT", "REP"))## TRAT REP N PROP sd se ci
## 1 Sin solarizar 1 6 28.7000000 16.7702117 6.8464103 17.5992578
## 2 Sin solarizar 2 6 29.5500000 19.6153766 8.0079440 20.5850753
## 3 Sin solarizar 3 6 17.0666667 14.1109414 5.7607677 14.8085248
## 4 Sin solarizar 4 6 15.4833333 9.5518410 3.8995228 10.0240424
## 5 Solarizado 1 1 6 1.6833333 2.2604572 0.9228278 2.3722043
## 6 Solarizado 1 2 6 2.8000000 2.6487733 1.0813572 2.7797171
## 7 Solarizado 1 3 6 2.3333333 2.5468935 1.0397649 2.6728008
## 8 Solarizado 1 4 6 2.7833333 2.0614720 0.8415924 2.1633822
## 9 Solarizado 2 1 6 0.9666667 0.9933110 0.4055175 1.0424159
## 10 Solarizado 2 2 6 0.6666667 0.9626353 0.3929942 1.0102238
## 11 Solarizado 2 3 6 1.1333333 0.9709102 0.3963724 1.0189078
## 12 Solarizado 2 4 6 0.4833333 0.5741661 0.2344023 0.6025503summarySE(datosT, measurevar = "PROP", groupvars = c("TRAT"))## TRAT N PROP sd se ci
## 1 Sin solarizar 24 22.7000 15.8556258 3.2365161 6.6952436
## 2 Solarizado 1 24 2.4000 2.2771072 0.4648126 0.9615381
## 3 Solarizado 2 24 0.8125 0.8714368 0.1778813 0.3679755# transformados
summarySE(datosT, measurevar = "LOPROP", groupvars = c("TRAT", "REP"))## TRAT REP N LOPROP sd se ci
## 1 Sin solarizar 1 6 1.4260827 0.2092257 0.08541605 0.2195690
## 2 Sin solarizar 2 6 1.4145227 0.2720823 0.11107713 0.2855329
## 3 Sin solarizar 3 6 1.1792996 0.2603267 0.10627795 0.2731962
## 4 Sin solarizar 4 6 1.1631582 0.2323220 0.09484508 0.2438070
## 5 Solarizado 1 1 6 0.3394807 0.2773663 0.11323432 0.2910781
## 6 Solarizado 1 2 6 0.4947727 0.2954746 0.12062699 0.3100815
## 7 Solarizado 1 3 6 0.4363980 0.2840033 0.11594386 0.2980432
## 8 Solarizado 1 4 6 0.5302139 0.2176584 0.08885865 0.2284184
## 9 Solarizado 2 1 6 0.2531705 0.2005901 0.08189058 0.2105064
## 10 Solarizado 2 2 6 0.1786077 0.1947729 0.07951571 0.2044016
## 11 Solarizado 2 3 6 0.2912923 0.2002437 0.08174915 0.2101429
## 12 Solarizado 2 4 6 0.1492592 0.1438290 0.05871795 0.1509393summarySE(datosT, measurevar = "LOPROP", groupvars = c("TRAT"))## TRAT N LOPROP sd se ci
## 1 Sin solarizar 24 1.2957658 0.2613633 0.05335056 0.11036404
## 2 Solarizado 1 24 0.4502163 0.2626273 0.05360857 0.11089777
## 3 Solarizado 2 24 0.2180824 0.1832201 0.03739964 0.07736704# #### gráficos iniciales
library(DescTools)Desc(datosT, plot = T) ## Describe datosT (data.frame):
## 1 - TRAT (factor)
##
## length n NAs unique levels dupes
## 7e+01 7e+01 0 3e+00 3e+00 y
## 100.0% 0.0%
##
## level freq perc cumfreq cumperc
## 1 Sin solarizar 2e+01 33.3% 2e+01 33.3%
## 2 Solarizado 1 2e+01 33.3% 5e+01 66.7%
## 3 Solarizado 2 2e+01 33.3% 7e+01 100.0%#### Gráficos con más detalles
library(lattice)
library(car)library(agricolae)with(datosT, xyplot(LOPROP ~ TRAT ))with(datosT, xyplot(LOPROP ~ TRAT | REP ))with(datosT, xyplot(LOPROP ~ TRAT | MUES))with(datosT, xyplot(LOPROP ~ MUES | TRAT))with(datosT, xyplot(LOPROP ~ REP | TRAT))with(datosT, xyplot(LOPROP ~ TRAT | REP , groups = MUES))with(datosT, xyplot(LOPROP ~ REP | TRAT, groups = MUES))with(datosT, xyplot(LOPROP ~ TRAT | MUES, groups = REP ))with(datosT, xyplot(LOPROP ~ MUES | TRAT, groups = REP ))with(datosT, xyplot(LOPROP ~ REP | MUES, groups = TRAT ))with(datosT, xyplot(LOPROP ~ MUES | REP , groups = TRAT ))with(datosT, xyplot(LOPROP ~ TRAT | REP , groups = MUES , aspect = "xy"))with(datosT, xyplot(LOPROP ~ REP | TRAT, groups = MUES , aspect = "xy"))with(datosT, xyplot(LOPROP ~ TRAT | MUES, groups = REP , aspect = "xy"))with(datosT, xyplot(LOPROP ~ MUES | TRAT, groups = REP , aspect = "xy"))with(datosT, xyplot(LOPROP ~ REP | MUES, groups = TRAT , aspect = "xy"))with(datosT, xyplot(LOPROP ~ MUES | REP , groups = TRAT , aspect = "xy"))with(datosT, xyplot(LOPROP ~ TRAT | REP , groups = MUES , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ REP | TRAT, groups = MUES , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ TRAT | MUES, groups = REP , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ MUES | TRAT, groups = REP , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ REP | MUES, groups = TRAT , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ MUES | REP , groups = TRAT , aspect = "xy", type = "o"))with(datosT, xyplot(LOPROP ~ TRAT | REP , groups = MUES , aspect = "xy", type = "a"))with(datosT, xyplot(LOPROP ~ REP | TRAT, groups = MUES , aspect = "xy", type = "a"))with(datosT, xyplot(LOPROP ~ TRAT | MUES, groups = REP , aspect = "xy", type = "a"))with(datosT, xyplot(LOPROP ~ MUES | TRAT, groups = REP , aspect = "xy", type = "a"))with(datosT, xyplot(LOPROP ~ REP | MUES, groups = TRAT , aspect = "xy", type = "a"))with(datosT, xyplot(LOPROP ~ MUES | REP , groups = TRAT , aspect = "xy", type = "a"))histogram(~LOPROP | interaction(TRAT, REP), data = datosT)histogram(~LOPROP | interaction(TRAT, MUES), data = datosT)library (ggplot2)# Histogramas + densidades (opción 1)
par(mfrow = c(1,2))
hist(PROP, prob = TRUE, col = "grey") # datos sin transformar
lines(density(PROP), col = "blue", lwd = 2)
lines(density(PROP , adjust = 2), lty = "dotted", col = "darkgreen", lwd = 2)
hist(LOPROP, prob = TRUE, col = "grey") # datos transformados
lines(density(LOPROP), col = "blue", lwd = 2)
lines(density(LOPROP , adjust = 2), lty = "dotted", col = "darkgreen", lwd = 2) # Histogramas + densidades (opción 2)
plot1 <- ggplot(data = datosT, aes(PROP)) + # datos no transformados
geom_histogram(aes(y = ..density..), breaks=seq(1, 75, by=4), col="grey", fill="green", alpha=.2) +
geom_density(col = 4) + labs(title = "Número de propágalos") +
labs(x = "V. dahliae por g de suelo (No.)", y = "Densidad")
plot2 <- ggplot(data = datosT, aes(LOPROP)) + # datos transformados
geom_histogram(aes(y = ..density..), breaks=seq(0, 2, by=.1), col="grey", fill="green", alpha=.2) +
geom_density(col = 4) + labs(title = "log10 de propágalos") +
labs(x = "V. dahliae por g de suelo (log10)", y = "Densidad")
# mostar gráficos juntos
library(gridExtra)grid.arrange(plot1, plot2, nrow=1, ncol=2)#### Gráficos Box and Whisker
# Box-plot (opción 1)
library(HH)BW1 <- bwplot(PROP ~ TRAT, data = datosT, scales=list(cex=1.5),
ylab=list("Número de propágalos", cex = 1.5),
xlab=list("Tratamientos de suelo",cex = 1.5)); BW1BW2 <- bwplot(LOPROP ~ TRAT, data = datosT, scales=list(cex=1.5),
ylab = list("log10, datos transformados", cex=1.5),
xlab=list("Tratamientos de suelo",cex=1.5)); BW2# Box-plot (opción 2)
#### Box-and-Whisker con puntos para inspeccionar valores atípicos
dotplot1 <- ggplot(data = datosT, aes(x = TRAT, y = PROP)) +
ggtitle("V. dahliae por g de suelo (No.)") +
xlab("Tratamientos de suelo") + ylab("Número de propágalos") +
geom_point(position = position_jitter(height = 0.2, width = 0.2), size = 2) +
geom_boxplot(alpha = 0.1)
dotplot2 <- ggplot(data = datosT, aes(x = TRAT, y = LOPROP)) +
ggtitle("V. dahliae por g de suelo (log10)") +
xlab("Tratamientos de suelo") + ylab("log10 de propágalos") +
geom_point(position = position_jitter(height = 0.2, width = 0.2), size = 2) +
geom_boxplot(alpha = 0.1)
# agrupando gráficos
grid.arrange(dotplot1, dotplot2, nrow=1, ncol=2)#### Box-plot TRAT*REP (opción 3)
par(mfrow = c(1,1))
boxplot(PROP ~ TRAT*REP, col = c("green", "red", "blue"), datosT) # sin transformarboxplot(LOPROP ~ TRAT*REP, col = c("green", "red", "blue"), datosT) # transformados#### ANOVA combinados
# Ejemplo de un ANOVA con un muestreo (pp. 169)
modelo1 <- aov(LOPROP ~ TRAT * REP + Error(TRAT + REP), datosT); summary(modelo1)## Error: TRAT
## Df Sum Sq Mean Sq
## TRAT 2 15.44 7.721
##
## Error: REP
## Df Sum Sq Mean Sq
## REP 3 0.07266 0.02422
##
## Error: Within
## Df Sum Sq Mean Sq F value Pr(>F)
## TRAT:REP 6 0.503 0.08385 1.5 0.194
## Residuals 60 3.354 0.05590library(nlme) # únicamente para determinar SS totalanova(glm(LOPROP ~ TRAT * REP, family = gaussian(link = identity)), test = "F")## Analysis of Deviance Table
##
## Model: gaussian, link: identity
##
## Response: LOPROP
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev F Pr(>F)
## NULL 71 19.3716
## TRAT 2 15.4419 69 3.9296 138.1261 <2e-16 ***
## REP 3 0.0727 66 3.8570 0.4333 0.7300
## TRAT:REP 6 0.5031 60 3.3539 1.5000 0.1937
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1#### El nuevo ANOVA (empleando diferentes códigos para el mismo propósito)
modelo2a <- aov(lm(LOPROP ~ TRAT + REP)); modelo2a## Call:
## aov(formula = lm(LOPROP ~ TRAT + REP))
##
## Terms:
## TRAT REP Residuals
## Sum of Squares 15.441932 0.072657 3.856973
## Deg. of Freedom 2 3 66
##
## Residual standard error: 0.2417416
## Estimated effects may be unbalancedmodelo2b <- aov(LOPROP ~ TRAT + REP); summary(modelo2b)## Df Sum Sq Mean Sq F value Pr(>F)
## TRAT 2 15.442 7.721 132.120 <2e-16 ***
## REP 3 0.073 0.024 0.414 0.743
## Residuals 66 3.857 0.058
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1modelo2c <- anova(glm(LOPROP ~ TRAT + REP, family = gaussian(link = identity)), test = "F");modelo2c## Analysis of Deviance Table
##
## Model: gaussian, link: identity
##
## Response: LOPROP
##
## Terms added sequentially (first to last)
##
##
## Df Deviance Resid. Df Resid. Dev F Pr(>F)
## NULL 71 19.3716
## TRAT 2 15.4419 69 3.9296 132.1201 <2e-16 ***
## REP 3 0.0727 66 3.8570 0.4144 0.7432
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1boxplot(LOPROP ~ TRAT,datosT)plot(modelo2a)library(car)
modelo3 <- lm(LOPROP ~ TRAT + REP)
Anova(modelo3, type = 'III')## Anova Table (Type III tests)
##
## Response: LOPROP
## Sum Sq Df F value Pr(>F)
## (Intercept) 20.7188 1 354.5374 <2e-16 ***
## TRAT 15.4419 2 132.1201 <2e-16 ***
## REP 0.0727 3 0.4144 0.7432
## Residuals 3.8570 66
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1#### Contrastes ortogonales
# coeficientes de TRAT
str(datosT)## 'data.frame': 72 obs. of 5 variables:
## $ TRAT : Factor w/ 3 levels "Sin solarizar",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ REP : Factor w/ 4 levels "1","2","3","4": 1 1 1 1 1 1 2 2 2 2 ...
## $ MUES : Factor w/ 6 levels "1","2","3","4",..: 1 2 3 4 5 6 1 2 3 4 ...
## $ PROP : num 18.4 19.8 23.6 60.2 34.5 15.7 28.3 65.4 23.1 34.5 ...
## $ LOPROP: num 1.29 1.32 1.39 1.79 1.55 ...c1 <- c(2,-1, -1)
c2 <- c(0, 1, -1)
# crear una matrix temporal
mat.temp <- rbind(constant = 1/3, c1, c2); mat.temp## [,1] [,2] [,3]
## constant 0.3333333 0.3333333 0.3333333
## c1 2.0000000 -1.0000000 -1.0000000
## c2 0.0000000 1.0000000 -1.0000000# obtiener matrix inversa
mat <- solve(mat.temp); mat## constant c1 c2
## [1,] 1 0.3333333 0.0
## [2,] 1 -0.1666667 0.5
## [3,] 1 -0.1666667 -0.5# eliminar la primera columna de la matrix
mat <- mat[ , -1]; mat## c1 c2
## [1,] 0.3333333 0.0
## [2,] -0.1666667 0.5
## [3,] -0.1666667 -0.5# cálculo de contrast (opción 1)
modelo5 <- lm(LOPROP ~ TRAT, data = datosT,
contrasts = list(TRAT = mat)); summary(modelo5)## Call:
## lm(formula = LOPROP ~ TRAT, data = datosT, contrasts = list(TRAT = mat))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.4149 -0.1741 -0.0525 0.1776 0.5264
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.65469 0.02812 23.28 < 2e-16 ***
## TRATc1 1.92323 0.11932 16.12 < 2e-16 ***
## TRATc2 0.23213 0.06889 3.37 0.00124 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.2386 on 69 degrees of freedom
## Multiple R-squared: 0.7971, Adjusted R-squared: 0.7913
## F-statistic: 135.6 on 2 and 69 DF, p-value: < 2.2e-16# cálculo de contrast (opción 2)
contrasts(TRAT) <- cbind(mat)
contraste <- aov(LOPROP ~ TRAT); summary.lm(contraste)## Call:
## aov(formula = LOPROP ~ TRAT)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.4149 -0.1741 -0.0525 0.1776 0.5264
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.65469 0.02812 23.28 < 2e-16 ***
## TRATc1 1.92323 0.11932 16.12 < 2e-16 ***
## TRATc2 0.23213 0.06889 3.37 0.00124 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.2386 on 69 degrees of freedom
## Multiple R-squared: 0.7971, Adjusted R-squared: 0.7913
## F-statistic: 135.6 on 2 and 69 DF, p-value: < 2.2e-16# medias de tratamientos solarizados
library(psych)describeBy(PROP, TRAT, mat = TRUE, digits = 2)## item group1 vars n mean sd median trimmed mad min max
## X11 1 Sin solarizar 1 24 22.70 15.86 16.45 20.27 10.16 6.6 65.4
## X12 2 Solarizado 1 1 24 2.40 2.28 1.35 2.13 1.26 0.2 7.3
## X13 3 Solarizado 2 1 24 0.81 0.87 0.50 0.70 0.52 0.0 2.8
## range skew kurtosis se
## X11 58.8 1.32 0.88 3.24
## X12 7.1 1.02 -0.45 0.46
## X13 2.8 1.16 0.05 0.18library(doBy)library(pastecs)summaryBy(PROP ~ TRAT, data = datosT, FUN = stat.desc)## TRAT PROP.nbr.val PROP.nbr.null PROP.nbr.na PROP.min PROP.max
## 1 Sin solarizar 24 0 0 6.6 65.4
## 2 Solarizado 1 24 0 0 0.2 7.3
## 3 Solarizado 2 24 4 0 0.0 2.8
## PROP.range PROP.sum PROP.median PROP.mean PROP.SE.mean PROP.CI.mean.0.95
## 1 58.8 544.8 16.45 22.7000 3.2365161 6.6952436
## 2 7.1 57.6 1.35 2.4000 0.4648126 0.9615381
## 3 2.8 19.5 0.50 0.8125 0.1778813 0.3679755
## PROP.var PROP.std.dev PROP.coef.var
## 1 251.4008696 15.8556258 0.6984857
## 2 5.1852174 2.2771072 0.9487947
## 3 0.7594022 0.8714368 1.0725377Códigos en SAS
/***********************************************************
EXPERIMENTACIÓN EN AGRICULTURA
Fernández
Escobar, R.; Trapero, A.; Domínguez, J. 2010
CAPÍTULO 13:
ANÁLISIS DE LA VARIANZA COMBINADO
(páginas
167 - 172)
************************************************************/
DATA
datos;
DO
TRAT = 0 TO 2;
DO REP
= 1 TO 4;
DO
MUES = 1 TO 6;
INPUT
PROP @@;
OUTPUT;
END;
END;
END;
LABEL TRAT = 'Tratamientos
del suelo'
REP = 'Repeticiones'
MUES = 'Muestras de suelo'
PROP = 'Propágalos de V. dahliae (g/suelo)';
DATALINES;
18.4 19.8 23.6 60.2 34.5 15.7
28.3 65.4 23.1 34.5 9.6 16.4
13.4 12.0 7.1
15.0 9.6 45.3
16.5 8.1 12.7
15.8 6.6 33.2
0.6 1.5
1.1 6.2 0.5 0.2
0.7 2.7
1.2 4.4 7.3 0.5
7.0 3.6
0.8 0.5 1.2 0.9
2.3 0.8
3.7 1.9 6.5 1.5
2.8 1.2
0.6 0.9 0.0 0.3
0.0 0.2
0.4 2.6 0.3 0.5
1.0 2.7
1.8 0.5 0.0 0.8
0.5 1.6
0.4 0.2 0.2 0.0
;
* Mostrar datos;
PROC PRINT DATA =
datos;
RUN;
* Etiquetar TRATAMIENTOS;
PROC FORMAT;
VALUE TRATCODIGO
0 = 'Sin
solarizar'
1 = 'Solarizado
1'
2 = 'Solarizado
2';
RUN;
DATA
datosID;
SET
datos;
FORMAT
TRAT TRATCODIGO. REP MUES PROP;
RUN;
* Datos con ID ;
PROC PRINT DATA =
datosID;
RUN;
* Transformación logarítmica (LOPROP);
DATA datosT;
SET
datosID;
LOPROP = log10(PROP+1);
LABEL = 'log(# propagalos)';
RUN;
* Datos + variable transformada;
PROC PRINT DATA =
datosT;
RUN;
* Estadísticas
descriptivas;
PROC MEANS MEAN STD STDERR;
VAR
PROP LOPROP;
CLASS
TRAT REP;
RUN;
* Opción 1: ANOVA bloques divididos;
PROC GLM DATA =
datosT;
CLASS
TRAT REP MUES;
MODEL
LOPROP = REP TRAT TRAT*REP / SS3;
TEST H
=TRAT REP E = TRAT*REP;
RUN;
* Opción 2: ANOVA
bloques divididos;
PROC MIXED DATA =
datosT METHOD = TYPE3;
CLASS
TRAT REP MUES;
MODEL
LOPROP = TRAT REP TRAT*REP*MUES/DDFM=SATTERTH;
RANDOM
REP*TRAT;
RUN;
PROC GLM;
CLASS
TRAT REP;
MODEL
LOPROP = TRAT REP;
MEANS TRAT;
CONTRAST 'No
solarizado vs. (Solarizado 1 + Solarizado 2)' TRAT 2 -1 -1;
CONTRAST 'Solarizado
1 vs. Solarizado 2' TRAT 0 1 -1;
RUN;
PROC MEANS MEAN STD;
VAR
PROP;
CLASS
TRAT;
RUN;
ODS HTML CLOSE;
------------------------------------------------------------
Archivos:
tabla 13.1.csv, capítulo 13a.R, summarySE.R, capítulo 13a.sas

No hay comentarios:
Publicar un comentario